今回は各種について2セットのゲノム(雌雄由来のそれぞれのハプロタイプ)を解析し、全染色体で合計215本分の“ギャップ(未解読領域)のない”配列が得られました。
(※より具体的には290本中215本がギャップなしで残りも平均1–6ギャップのみという高精度の配列が得られました)
この精度はヒトゲノムの最新リファレンス配列に匹敵し、過去の類人猿ゲノムのドラフト配列で問題となっていた欠損やエラーが大幅に解消されています。
ヒトと類人猿を比較する際、これまではヒト側の配列品質が高いために起こるバイアスも指摘されていましたが、今回のデータセットではヒトも他の類人猿も同等の品質で比較できるようになりました。
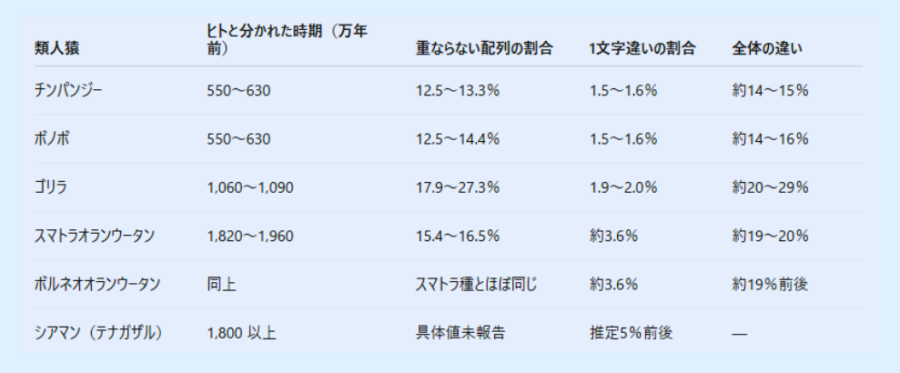
例えば今回、新たに得られたゲノム配列を用いてヒトとチンパンジーを比較したところ、これまで「揃わないため」に無視されていた領域を含めると、両者のゲノムの違いは最大で15%にも達することが示されました。
従来の“一塩基置換”レベルでの違い(約1~1.5%程度)に加えて、どちらか一方にしか存在しない配列(大規模な挿入・欠失や重複の差異)が全ゲノムの1割以上を占めていたのです。
言い換えれば、ヒトとチンパンジーのゲノムを丁寧に重ね合わせていくと、約8分の1(12.5~13.3%)もの領域で対応する配列同士がずれたり欠落したりして、もはや整合しないことが分かったのです。
これはセントロメアやサブテロメア(染色体の端近く)などに存在する急速に進化した領域で特に顕著でした。
また、従来「1%」と言われた一塩基レベルの違いについても、厳密に測れば約1.5%ほどあることが示されています。
以上を総合すると、ヒトとチンパンジーのゲノムの最大15%(14~14.9%)が何らかの形で相違しているということになります。
では、その「見えてきた差異」とは具体的にどのようなものだったのでしょうか。











































